Hinweise
Eine R-Nutzerin, die bereits vorher einen R-Kurs belegt hat, bewertete dieses Tutorial insgesamt mit einer Schwierigkeit von 5 (0=sehr leicht, 10=sehr schwer). Sie brauchte für dieses Tutorial ungefähr 1h 45min (ohne die letzte Übungsaufgabe). Klicke auf “Nächstes Kapitel” und es geht los.
Einführung
Wie am Ende des zweiten Tages versprochen geht es nun mit richtiger Methodenlehre los. Wir werden heute und in den restlichen Sitzungen, Schritt für Schritt, alle statistischen Themen der Vorlesungen Methodenlehre I und II abhandeln. Los geht es mit deskriptiver Statistik. Als Datensatz benutzen wir eine Vorlesungsbefragung aus Methodenlehre I. Schauen wir uns die Daten mal grob an:
data <- readRDS("data/students.rds")
head(data)library(psych)
describe(data)Tabellen
Machen wir erst mal eine einfache Tabelle für die Punkte im Matheabitur:
table(data$mathe)##
## 0 1 2 4 5 6 7 8 9 10 11 12 13 14 15
## 1 1 3 1 2 4 6 4 15 12 15 16 16 8 5Nun einige Erweiterungen. Tabelle nach Gruppenvariable:
table(data$mathe, data$geschlecht)##
## divers männlich weiblich
## 0 1 0 0
## 1 0 1 0
## 2 0 1 2
## 4 0 0 1
## 5 0 0 2
## 6 0 1 3
## 7 0 0 6
## 8 0 0 4
## 9 0 5 10
## 10 1 3 8
## 11 0 2 13
## 12 0 3 13
## 13 0 4 12
## 14 0 1 7
## 15 0 2 3Relative Häufigkeiten gerundet:
round(proportions(table(data$mathe, data$geschlecht), 2), 2)##
## divers männlich weiblich
## 0 0.50 0.00 0.00
## 1 0.00 0.04 0.00
## 2 0.00 0.04 0.02
## 4 0.00 0.00 0.01
## 5 0.00 0.00 0.02
## 6 0.00 0.04 0.04
## 7 0.00 0.00 0.07
## 8 0.00 0.00 0.05
## 9 0.00 0.22 0.12
## 10 0.50 0.13 0.10
## 11 0.00 0.09 0.15
## 12 0.00 0.13 0.15
## 13 0.00 0.17 0.14
## 14 0.00 0.04 0.08
## 15 0.00 0.09 0.04Ein kleiner Check: Beträgt die Summe dieser Tabelle 100%?
sum(proportions(table(data$mathe, data$geschlecht)))## [1] 1proportions selbst nimmt als Argument eine normale Tabelle, die wir vorher über table erzeugen. Das mag für Rohdaten etwas umständlich erscheinen, ist aber ganz praktisch, wenn wir von irgendwoher eine fertige Tabelle bekommen und dort die relativen Anteile wissen wollen:
irgendeine_tabelle <- matrix(c(10, 30, 14, 28), nrow = 2)
irgendeine_tabelle## [,1] [,2]
## [1,] 10 14
## [2,] 30 28round(proportions(irgendeine_tabelle), 2)## [,1] [,2]
## [1,] 0.12 0.17
## [2,] 0.37 0.34Relative Häufigkeiten nach Spalten (gerundet):
round(proportions(table(data$mathe, data$geschlecht), margin = 2), 2)##
## divers männlich weiblich
## 0 0.50 0.00 0.00
## 1 0.00 0.04 0.00
## 2 0.00 0.04 0.02
## 4 0.00 0.00 0.01
## 5 0.00 0.00 0.02
## 6 0.00 0.04 0.04
## 7 0.00 0.00 0.07
## 8 0.00 0.00 0.05
## 9 0.00 0.22 0.12
## 10 0.50 0.13 0.10
## 11 0.00 0.09 0.15
## 12 0.00 0.13 0.15
## 13 0.00 0.17 0.14
## 14 0.00 0.04 0.08
## 15 0.00 0.09 0.04margin bedeutet Rand, es kommt also an den Rand noch eine Zeile/Spalte hinzu, die die Daten zusammenfasst (in diesem Fall Randsummen oder Randanteile). Bei einer ANOVA-Tabelle machen wir so etwas ähnliches, benutzen jedoch Mittelwerte. Die 2 bei margin bedeutet Spalte (1 wäre die Zeile).
Okay. Dann probier Du es nun mal. Zeig mir die Häufigkeitsverteilung der Abiturnoten aufgeteilt nach dem Geschlecht:
table(data$abi, data$geschlecht)Und nun das Gleiche mit relativen Häufigkeiten:
proportions(table(data$abi, data$geschlecht))Gib mir den relativen Anteil der Punkte in Deutsch nach dem Geschlecht aus. Die Summe über das Geschlecht soll in diesem Fall 100% ergeben. Zum Beispiel: für 6 Punkte, männlich 25%, weiblich 75%.
# Schau Dir die Befehle von oben an. Es gibt ein sehr ähnliches Beispiel.# Benutze das Argument margin = 1 und rowSums, wenn Du die Randsummen nach der
# Zeile bilden willst.proportions(table(data$deutsch, data$geschlecht), margin = 1)
# Summe 100%?
rowSums(proportions(table(data$deutsch, data$geschlecht), margin = 1))Man kann natürlich noch kompliziertere Tabellen mit mehr Variablen erzeugen, aber das wird schnell unübersichtlich und wird nur selten benötigt. Für eine Zusammenfassung von mehr als drei Variablen bieten sich eher Lage- und Streuungsmaße an.
Lage- und Streuungsmaße
Die bekanntesten Lage- und Streuungsmaße kennst Du bereits aus Tag 2:
Mittelwert
mean(data$abi)## [1] 1.866972Median
median(data$abi)## [1] 1.6Standardabweichung
sd(data$abi)## [1] 0.5647115Welche Maße sind sonst noch relevant in der Methodenlehre? Berechne mir vom Alter die Varianz, die Range, den mittleren absoluten Abstand, die Quantile 30% und 60%, sowie den Interquartilsabstand.
var(data$alter)
range(data$alter)
mad(data$alter)
quantile(data$alter, c(0.3, 0.6))
IQR(data$alter)Uns fehlt nur noch der Modus. Wie Du schon aus Tag 1 weißt, gibt es keine Funktion in R für den Modus (zumindest ohne Zusatzpakete). Wir werden uns jetzt eine eigene Funktion dafür schreiben. Das ist ein relativ fortgeschrittenes Thema, aber wir sind ja auch schon bei Tag 3. Konzentrier Dich gut, dann hast Du bald die Möglichkeit R so zu erweitern, wie Du willst.
Exkurs: Eigene Funktionen schreiben
Wir überlegen uns zunächst was die Funktion Modus leisten soll, welche Eingaben sie braucht und welche Ausgabe sie produziert.
Der Modus ist der häufigste Wert, also könnten wir die Funktion table benutzen
table(data$alter)##
## 17 18 19 20 21 22 23 24 25 26 27 28 30 33 36 43
## 2 18 40 20 4 8 4 1 3 1 2 1 1 1 2 1und von dort den maximalen Wert auslesen:
max(table(data$alter))## [1] 40Das ist allerdings nicht der Wert selbst, sondern wie oft er vorkommt. Wir müssen jetzt also noch selektieren:
tab_alter <- table(data$alter)
max_frequency <- max(table(data$alter))
tab_alter[tab_alter == max_frequency]## 19
## 40Das scheint zu funktionieren. Als Output möchten wir nur den Wert und nicht die Häufigkeit:
tab_alter <- table(data$alter)
max_frequency <- max(table(data$alter))
names(tab_alter[tab_alter == max_frequency])## [1] "19"Das ganze noch als numerischen Wert:
tab_alter <- table(data$alter)
max_frequency <- max(table(data$alter))
as.numeric(names(tab_alter[tab_alter == max_frequency]))## [1] 19Das sollte für unsere Zwecke reichen. Wie Du siehst, braucht man doch einige Operationen um so etwas simples wie den Modus auszurechnen. Wir möchten das Ganze nun in eine Funktion packen, denn so viele Operationen auszuführen ist sehr umständlich. Als Eingabe haben wir nur den Vektor von dem wir gerne den Modus hätten, die Ausgabe ist ein Skalar.
modus <- function(x) {
tab <- table(x)
max_frequency <- max(table(x))
modus <- as.numeric(names(tab[tab == max_frequency]))
return(modus)
}
modus(data$alter)## [1] 19Wir erzeugen also eine Funktion indem wir einer Variablen den Wert function zuweisen. Die Funktion function hat eine spezielle Syntax. Sie beginnt mit Klammern (), die die Parameter der Funktion enthalten, in diesem Fall einfach einen Vektor mit dem Namen x. Du könntest auch eine andere Bezeichnung verwenden, wichtig ist aber, dass immer dort wo diese Variable gebraucht wird, Du den gleichen Namen benutzt (siehe Zeile 2 und 3, dort steht x). Probier es mal aus. Zunächst falsch: ändere table(x) zu table(y) und führe den Code nochmal aus.
modus <- function(x) {
tab <- table(____)
max_frequency <- max(table(x))
modus <- as.numeric(names(tab[tab == max_frequency]))
return(modus)
}
modus(data$alter)Und jetzt ändere mal alle Vorkommnisse von x zu y in der Funktion. Klappt es nun?
modus <- function(____) {
tab <- table(____)
max_frequency <- max(table(____))
modus <- as.numeric(names(tab[tab == max_frequency]))
return(modus)
}
modus(data$alter)Nach den Parametern kommen geschweifte Klammern {}, die die Funktion definieren. Das sind die Befehle, die wir uns vorher überlegt haben, jedoch mit der Variable x statt data$alter, denn die Funktion soll ja allgemein gültig sein. Abschließend benutzen wir die Anweisung return, die den finalen Wert der Funktion zurückgibt. Man könnte auch einfach modus schreiben statt return(modus), da die letzte Zeile bei R immer als Rückgabewert behandelt wird. Es ist aber sauberer return zu schreiben. Trotzdem, probier mal aus return zu entfernen und lass als letzte Zeile nur modus stehen. Das sollte zum gleichen Ergebnis führen.
modus <- function(x) {
tab <- table(x)
max_frequency <- max(table(x))
modus <- as.numeric(names(tab[tab == max_frequency]))
____
}
modus(data$alter)Der Aufruf der gesamten Funktion läuft über den Namen, den wir der Funktion gegeben haben (modus) und die Parameter, die die Funktion hat, in diesem Fall nur einer (x). Wir können den Parameter beim Aufruf benennen, was etwas sauberer ist:
modus(x = data$alter)## [1] 19Falls wir das nicht tun, geht R davon aus, dass die übergebenen Parameter der Reihenfolge der Funktionsparameter entsprechen. Da wir hier nur einen Parameter haben, spielt das keine große Rolle. Probier es mal aus, entferne x = und schreib nur modus(data$alter).
modus(x = data$alter)Funktionen wirken am Anfang sehr kompliziert. Im Grunde sind sie es aber nicht, denn Du musst nur die Syntax beachten. Jede Funktion hat einen Namen, sie hat Parameter, einen Rückgabewert und eine Definition.
Übung macht den Meister. Schreib eine eigene Funktion für die Berechnung des Mittelwerts. Benutze dafür die Funktionen sum und length. Berechne anschließend den Mittelwert für das Alter der Probanden, vergleiche ob Dein Ergebnis mit der Funktion mean übereinstimmt.
mw <- function(x){
# und hier nun Dein Code
}mw <- function(x){
return(sum(x) / length(x))
}
mw(data$alter) == mean(data$alter)Man hat hier also sehr viele Möglichkeiten, aber am saubersten ist es mit return zu arbeiten. Interessant ist mw <- mean, was einfach nur einen Alias für mean erzeugt. Wenn Dir also eine Funktion in R zu lang ist, kannst Du so ein Kürzel definieren.
Halte mal kurz inne. Du hast gerade eine eigene Funktion in R geschrieben. Du hast die Pforte der Allmächtigkeit geöffnet. Von nun an sagst Du R wie es mit Dir sprechen soll.
mach_mir_mal_den_mittelwert <- function(x) {
return(mean(x))
}
mach_mir_mal_den_mittelwert(data$alter)## [1] 20.68807und_jetzt_noch_die_varianz <- function(x){
return(var(x))
}
und_jetzt_noch_die_varianz(data$alter)## [1] 16.1981Respekt! Wenn Funktionen schreiben mal nicht einen Heidenspaß macht…
Nun aber wieder zum Ernst des Lebens. Was hatten wir noch in Methodenlehre 1? Na klar, Abbildungen!
Einfache Abbildungen
Boxplot gefällig?
boxplot(data$abi)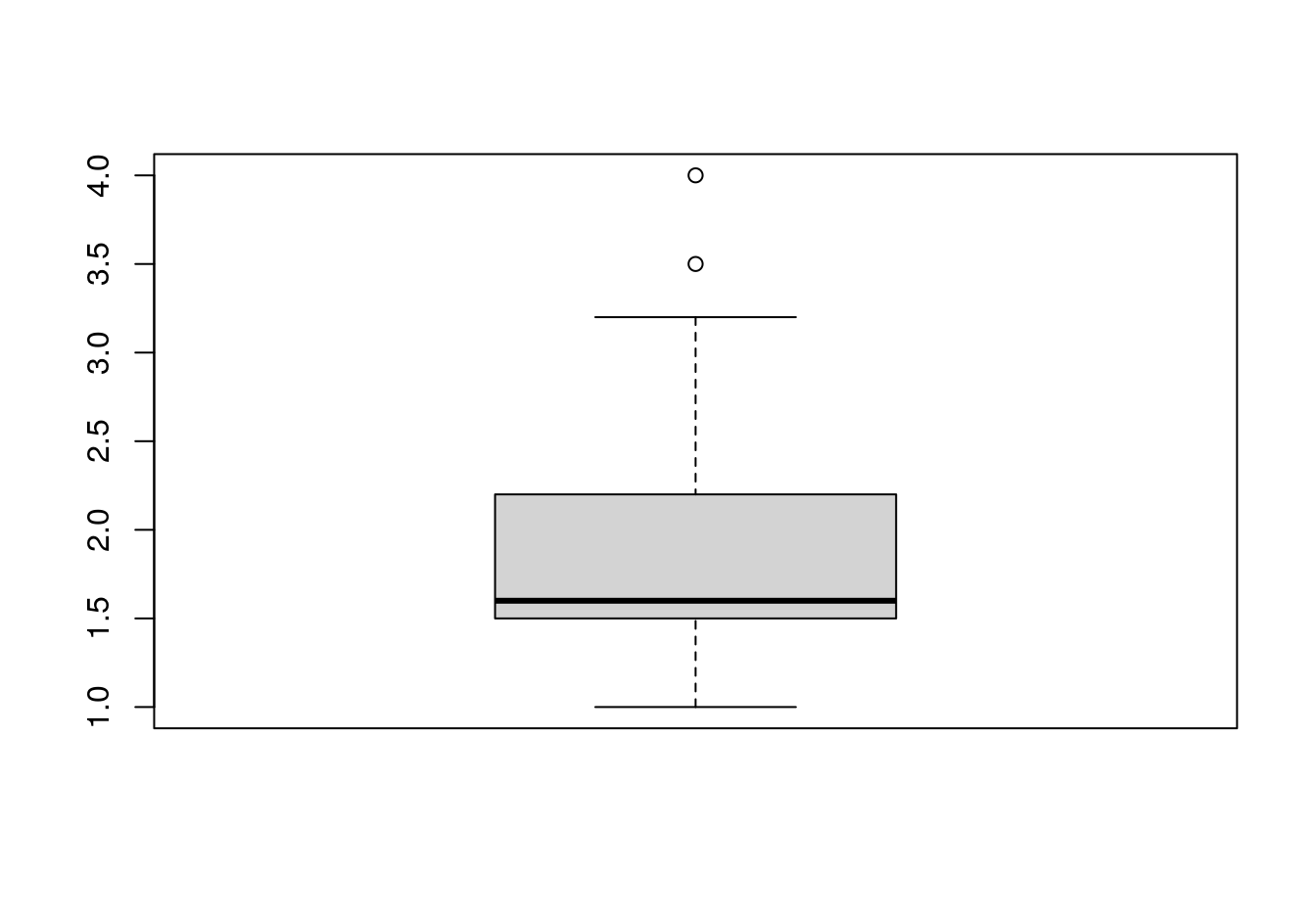
Oder doch lieber ein Stamm-Blatt-Diagramm?
stem(data$abi)##
## The decimal point is 1 digit(s) to the left of the |
##
## 10 | 0000
## 12 | 000
## 14 | 000000000000000000000000000
## 16 | 00000000000000000000000000000000000000
## 18 | 0000
## 20 | 0000
## 22 | 00000
## 24 | 0000000
## 26 | 000000
## 28 | 000000
## 30 | 00
## 32 | 0
## 34 | 0
## 36 |
## 38 |
## 40 | 0Boxplot mit Gruppierung:
boxplot(data$abi ~ data$geschlecht)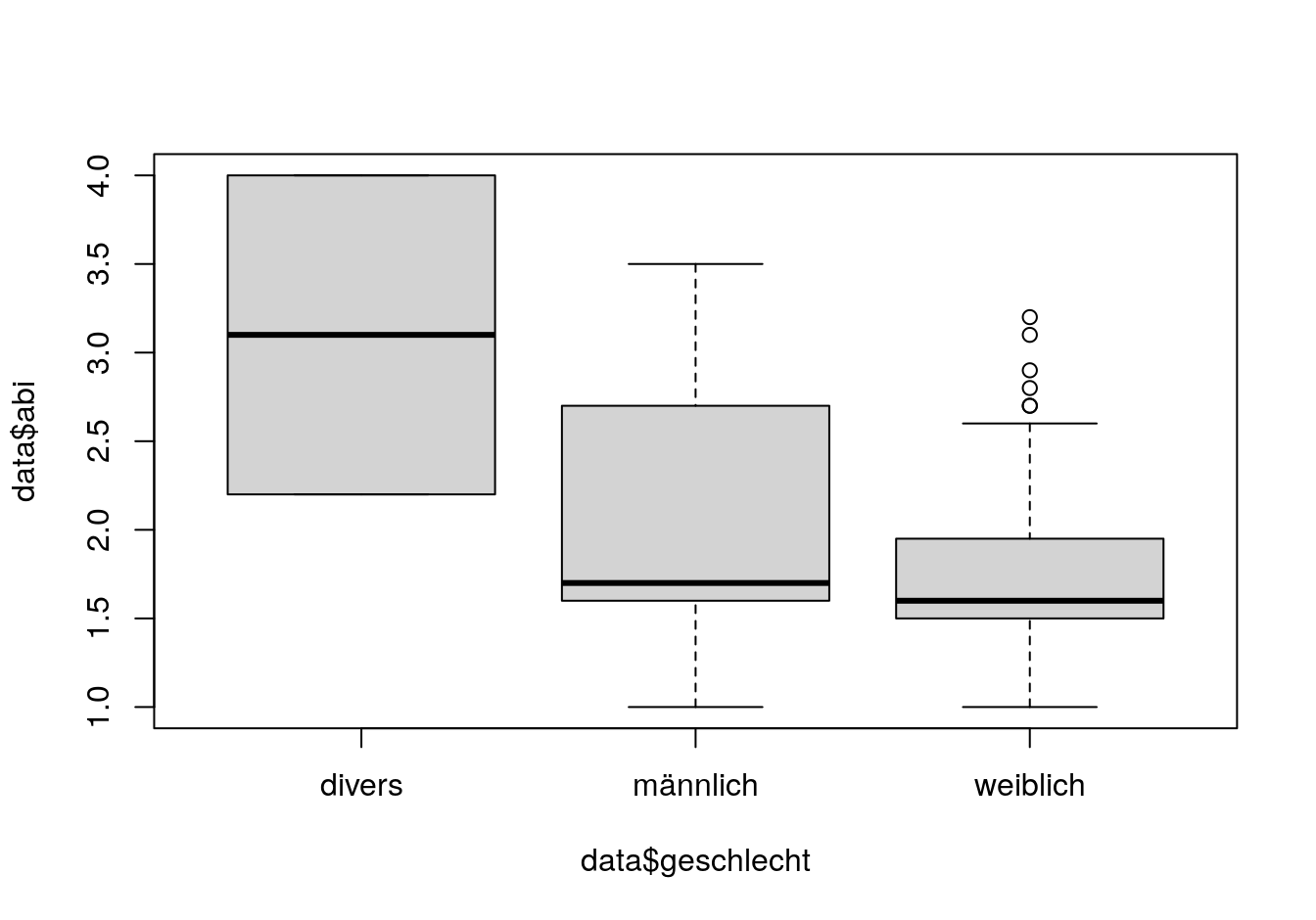
Hier benutzen wir die Tilde ~. Das ist ein spezielles Symbol in R, wir werden es uns später nochmal genauer anschauen. Merk Dir einfach, dass die Syntax der Idee folgt AV ~ UV1 + UV2 + ... + UVn (links steht die AV, rechts die UVs, getrennt durch die Tilde) – bitte frag mich jetzt nicht wo die Tilde auf der Tastatur ist. Ein echter Programmierer weiß das intuitiv!
Histogramm:
hist(data$abi)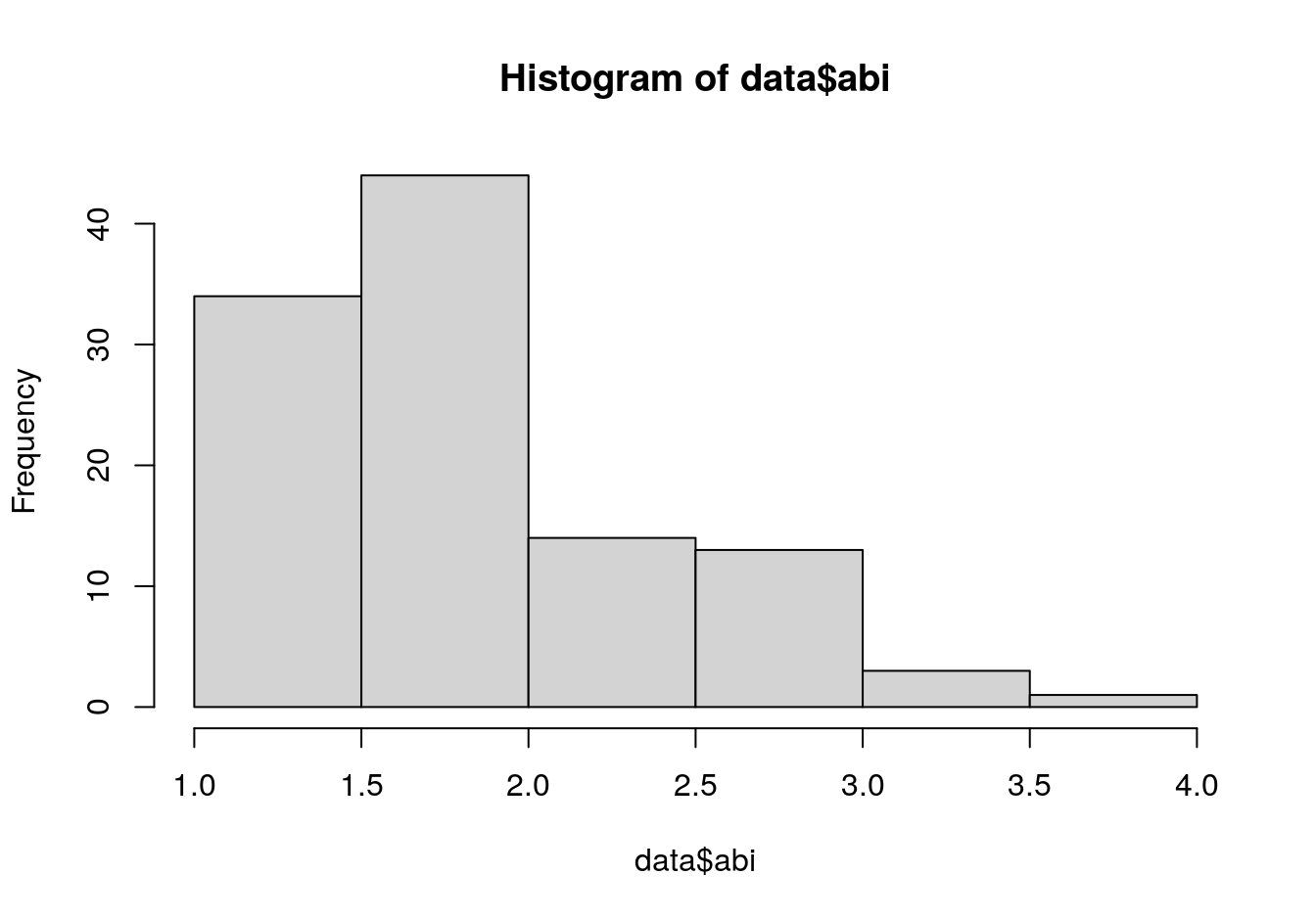
Streudiagramm
plot(data$mathe, data$deutsch)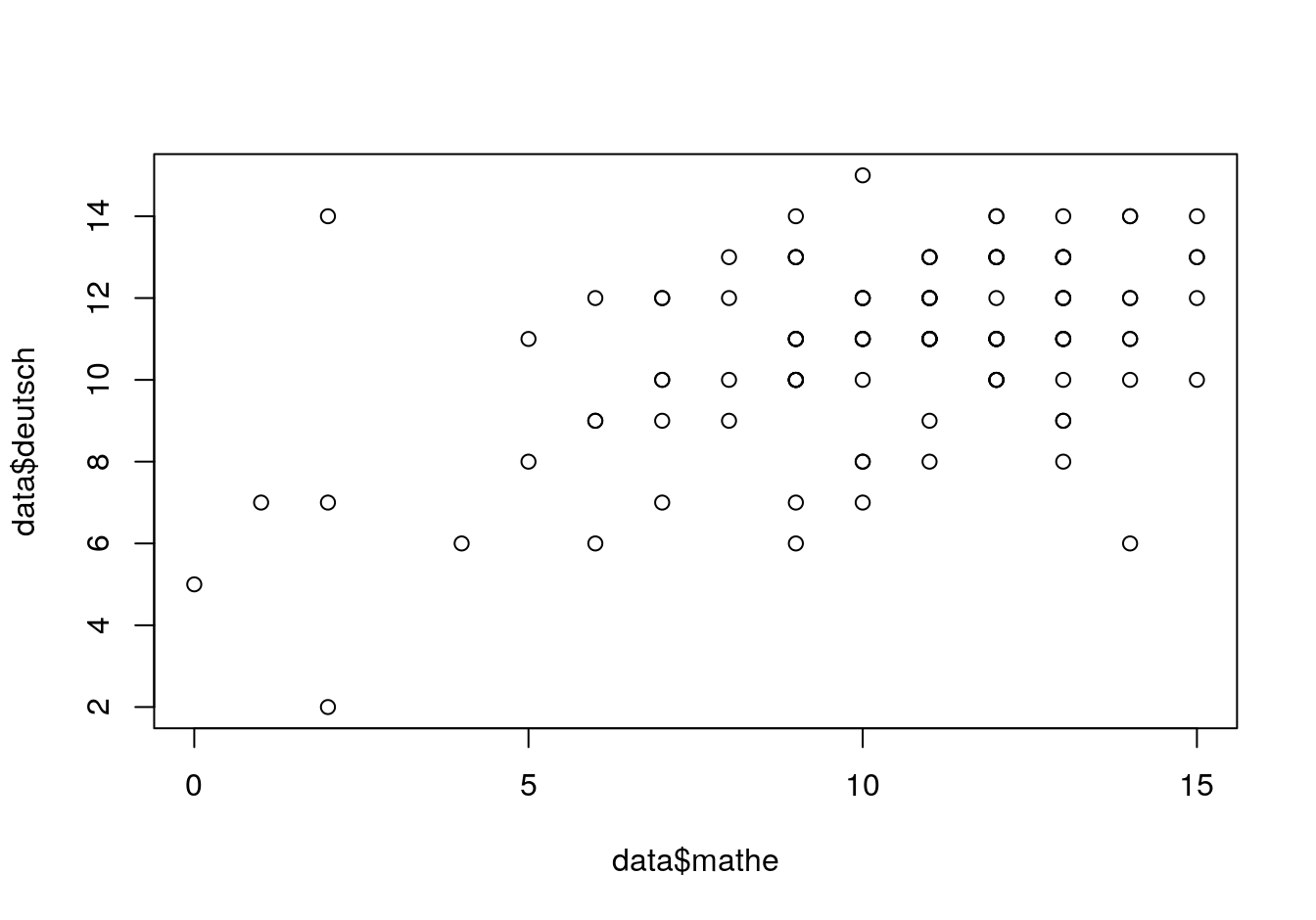
und dazu noch die Korrelation als Statistik:
cor(data$mathe, data$deutsch)## [1] 0.4798725Viele behaupten ja, dass man entweder gut in Deutsch oder Mathe ist. Dies ist offensichtlich nicht der Fall, denn die Korrelation zwischen beiden ist sehr hoch (wenn man den Messfehler berücksichtigt wäre die Korrelation noch deutlich höher). Aus Perspektive der meisten Intelligenztheorien ist dies ein zu erwartendes Resultat.
Unschön bei der Abbildung ist das Problem des Overplottings. Man sieht nicht, wenn mehrere Punkte aufeinanderliegen. Das lässt sich mit einem Sonnenblumendiagramm beheben:
sunflowerplot(data$mathe, data$deutsch)Oder mit Transparenz:
plot(data$mathe, data$deutsch, pch = 20, col = rgb(red = 0, green = 0, blue = 1,
alpha = 0.15))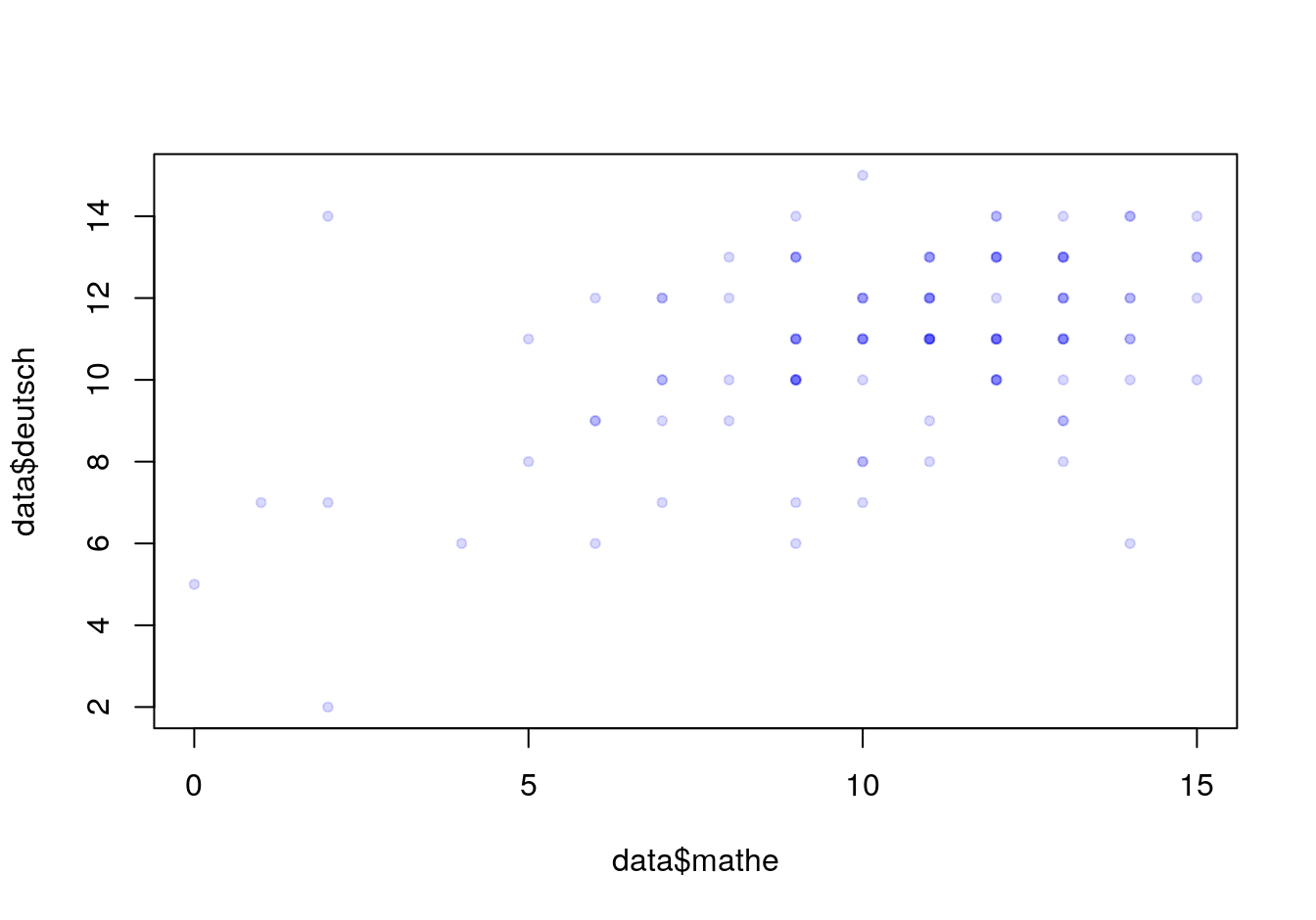
Was passiert hier? Wir machen einen Plot, der als Argument 1 die \(x\)-Achse aufnimmt, als Argument 2 die \(y\)-Achse. Dann übergeben wir noch pch, steht für plotting character. (Probier mal andere Zahlen aus!) Und schließlich noch über col die Farbe. Hierfür verwenden wir wiederum eine andere Funktion, rgb, und geben dort die Transparenz an. Probier für alpha andere Werte aus (zwischen 0 und 1).
Plots, die man heutzutage in den meisten Publikationen sieht, zeigen Mittelwerte und Standardfehler. Hierfür können wir das Hilfspaket psych benutzen. Dort gibt es die Funktion error.bars:
error.bars(data[, c("mathe", "deutsch")], eyes = FALSE)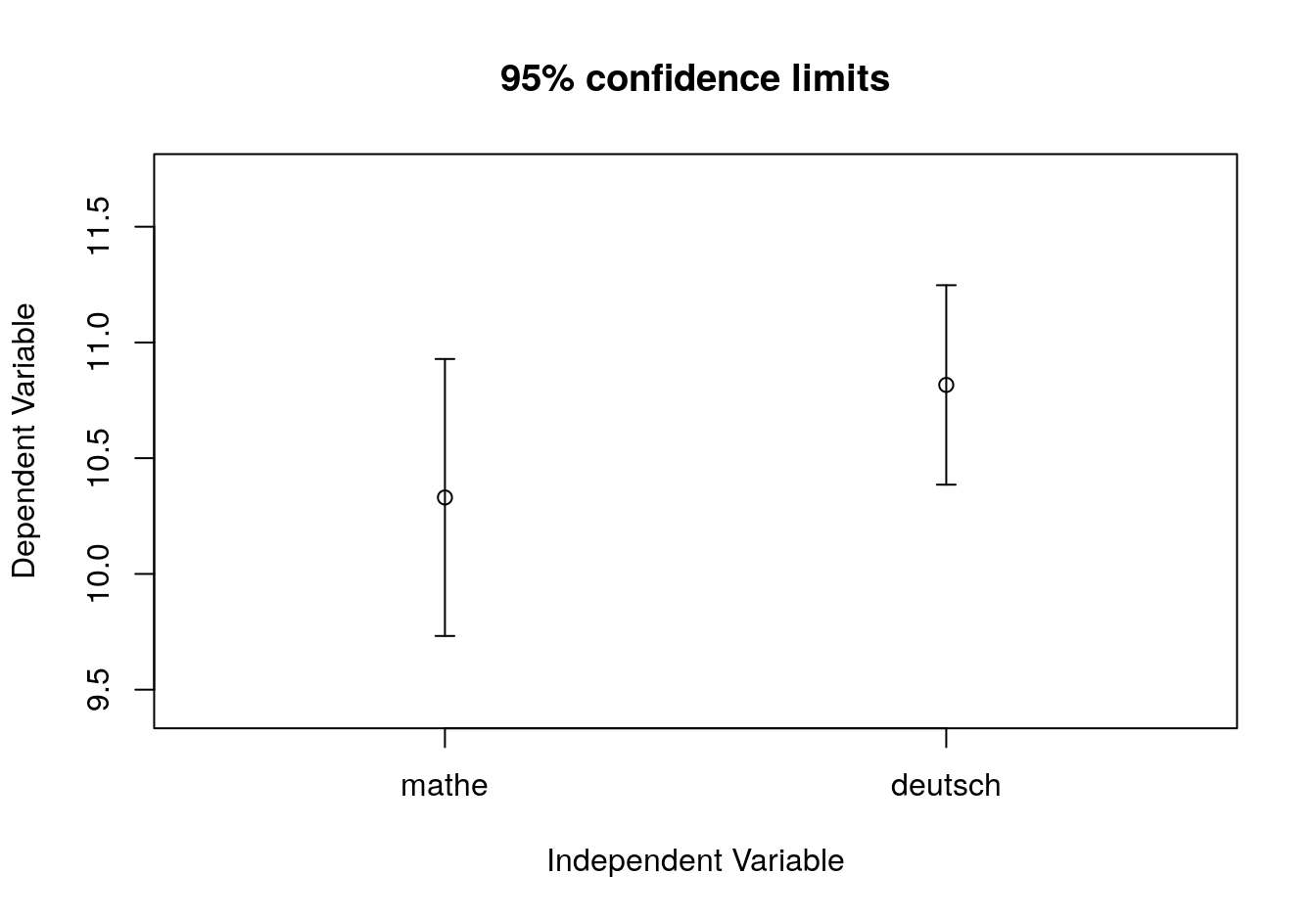
Das eyes-Argument kannst Du direkt mal ausprobieren. Setze es auf TRUE.
Die Abbildungen sehen schon ganz nett aus, aber noch bessere bekommt man mit dem Paket ggplot2 hin. Wenn Du also nach diesem Kurs noch mehr willst, empfehle ich Dir dieses Paket zu studieren.
Jetzt bist Du erst mal dran. Ich befördere Dich zum Hilfswissenschaftler. Deine erste Aufgabe: Mach mir ein paar schöne Plots!
Bitte ein Boxplot der Lebenszufriedenheit (Variable lebenszufriedenheit):
boxplot(data$lebenszufriedenheit)Nun der gleiche Boxplot, aber gruppiert nach Vegetarier und Meditation:
# Syntax für boxplot ist: AV ~ UV1 + UV2boxplot(data$lebenszufriedenheit ~ data$vegetarier + data$meditation)Nun bitte noch ein Stamm-Blatt-Diagramm für die Körpergröße (Variable groesse):
stem(data$groesse)Da hat sich wohl jemand einen Spaß erlaubt.
Jetzt noch ein Histogramm für Körpergröße:
hist(data$groesse)Nun einen Scatterplot der Abi-Note gegen die Lebenszufriedenheit (beim Abi sind niedrige Werte gut, bei der Lebenszufriedenheit sind hohe Werte gut):
plot(data$abi ~ data$lebenszufriedenheit)Beseitige das Problem des Overplottings (in der letzten Abbildung) mit Hilfe eines Sonnenblumendiagramms oder der Einführung von Transparenz.
sunflowerplot(data$abi ~ data$lebenszufriedenheit)
# oder
plot(data$abi ~ data$lebenszufriedenheit, pch = 20,
col = rgb(red = 0, green = 0, blue = 1, alpha = 0.15))Gut, nun noch die Korrelation zwischen Lebenszufriedenheit und der Abiturnote:
cor(data$abi, data$lebenszufriedenheit)
# für die nächste Aufgabe musst Du dann die Korrelation quadrieren, damit
# Du auf die Varianzaufklärung kommst! r²Zum Abschluss hätte ich gerne noch die Mittelwerte und das 68%-Konfidenzintervall für das Mögen von Hunden und Katzen (Variablen hunde_m und katzen_m). Nutze hier für die Funktion error.bars aus dem psych Paket und den Parameter alpha (Achtung: Alpha ist nicht das Konfidenzniveau!).
error.bars(data[, c("hunde_m", "katzen_m")], alpha = .32, eyes = FALSE)Sehr gute Arbeit! Deine Bezahlung erhältst Du natürlich nicht in € sondern in Rfahrungspunkten.
Viel mehr wollen wir heute gar nicht machen, denn gute Abbildungen zu erstellen, ist schon ziemlich aufwändig. Falls Du das nicht so siehst, kommen in den Übungsaufgaben für Tag 3 noch einige Challenges auf Dich zu.
Übungsaufgaben Tag 3
Programmiere eine eigene Funktion, die eine Variable \(z\)-standardisiert. Wende Sie anschließend auf die Mathe- und Deutschnote an. Multipliziere schließlich alle \(z\)-Werte der Mathe- und Deutschpunkte, summiere alle Wert auf und teile durch die Stichprobengröße (109).
zstandardisere <- function(x){
(x - mean(x)) / sd(x)
}zstandardisiere <- function(x){
(x - mean(x)) / sd(x)
}
sum(zstandardisiere(data$mathe) * zstandardisiere(data$deutsch)) / 109Erstelle eine Kreuztabelle zu den Variablen Studium und Geschlecht. Die Tabelle soll die relative Häufigkeit angeben und auf 2 Dezimalstellen gerundet sein. Falls Du nicht weißt wie man rundet, finde es durch eine Websuche heraus.
round(proportions(table(data$studium, data$geschlecht)), 2)Die Varianz die R über var berechnet ist die geschätzte Populationsvarianz (im Nenner steht \(n-1\)). Schreib eine Funktion, die die Stichprobenvarianz berechnet (im Nenner steht \(n\)). Vergleiche anschließend die absolute Abweichung der beiden Funktionen für die Variable Alter.
var_stichprobe <- function(x){
sum((x - mean(x))^2) / length(x)
}
var(data$alter) - var_stichprobe(data$alter)Spielen wir nun ein Spiel. Ich zeige Dir eine Abbildung und Du baust sie nach. Du musst in diesem Fall selbst nachprüfen ob Du alles richtig gemacht hast. Achte auf die Details!
Abbildung 1
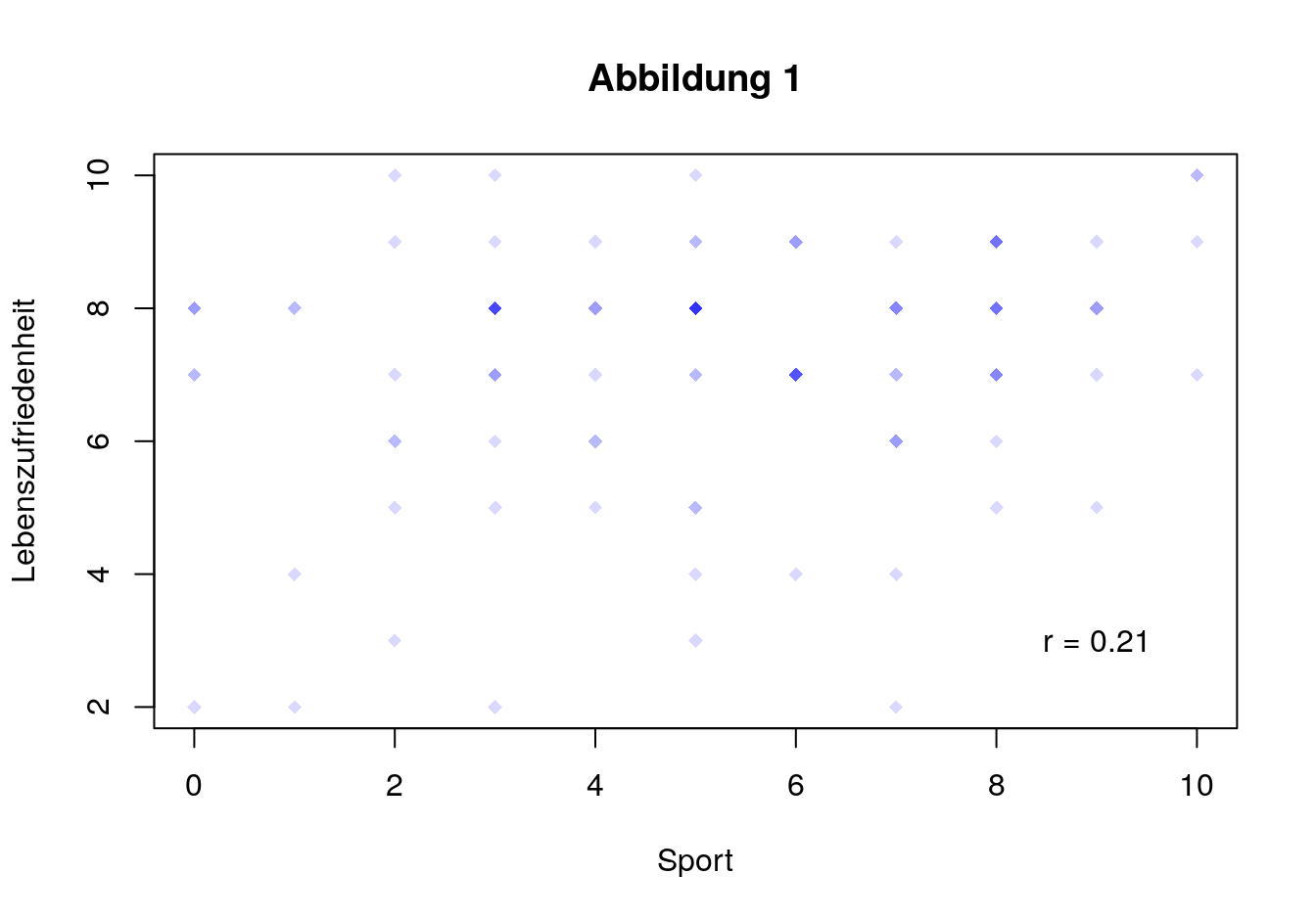
Nun Du. Wenn Du nicht weiter weist, dann mache eine Websuche und finde die notwendigen Parameter.
plot(data$sport, data$lebenszufriedenheit, pch = 18, col = rgb(0, 0, 1, 0.15),
xlab = "Sport", ylab = "Lebenszufriedenheit", main = "Abbildung 1")
text(9, 3, "r = 0.21")Abbildung 2
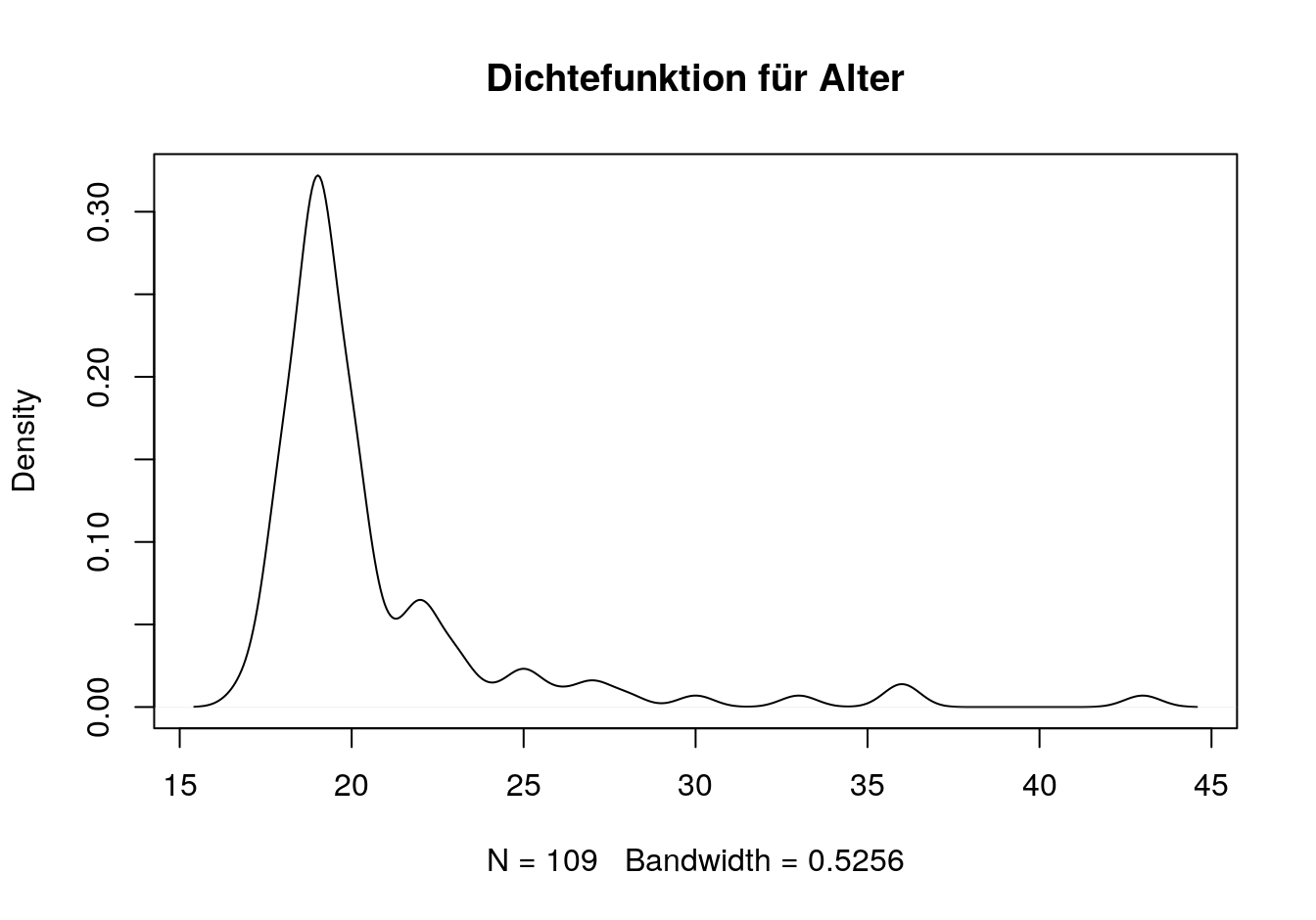
Nun Du. Wenn Du nicht weiter kommst, mache eine Websuche für “kernel density plot in R”.
plot(density(data$alter), main = "Dichtefunktion für Alter")Abbildung 3
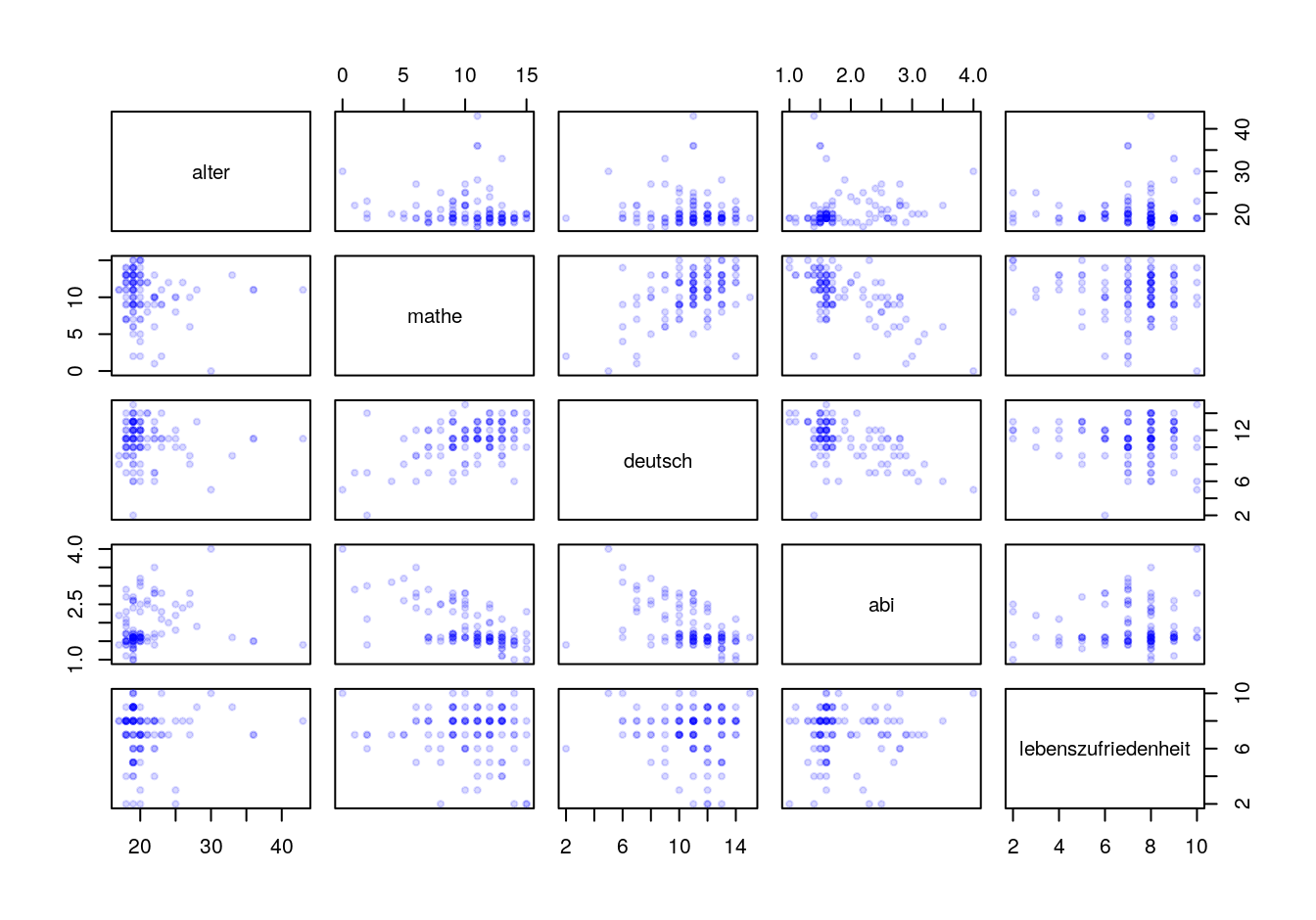
Nun Du. Wenn Du nicht weiter kommst, mache eine Websuche für “scatter plot matrices in R”.
pairs(data[,4:8], pch = 20, col = rgb(0, 0, 1, alpha = .15))Abbildung 4
Okay, und jetzt mal eine richtige Herausforderung!

Hierfür bekommst Du aber keinen Tipp, außer, dass es wirklich geht und dass es mehrere Lösungen gibt. Da man in diesem Tutorial keine externen Pakete (für alternative Lösungen) installieren kann, darfst Du diese Aufgabe auch lokal lösen (außerhalb des Browsers), schick mir dann zur Prüfung einfach den R-Code per Mail an halloffame at rlernen.de
# schau nochmal, was so im data-Ordner liegt und finde heraus welche Pakete
# geladen sind, dann siehst Du nämlich welches Paket ich benutzt habe...# So einfach wird das nicht! Aber wenn Du die Lösung wirklich hast, kannst Du
# sie mir schicken (halloffame at rlernen.de) und wenn sie stimmt, liste ich Dich
# in der Hall of Fame!Bisher haben die letzte Aufgabe nur zwei Personen geschafft
HALL OF FAME: Aline Mangold, Victoria Nöther